En los últimos años, debido a la alta demanda de los productos adicionados con probióticos y los múltiples beneficios nutricionales y terapéuticos asociados, la investigación sobre estos microorganismos ha progresado considerablemente, se han realizado avances notables en su selección y caracterización. De manera general, diversas entidades como la Organización Mundial de la Salud (OMS) y la Organización de las Naciones Unidas para la agricultura y la alimentación (FAO) recomiendan que se declare en la etiqueta del producto, tanto la especie como la cantidad de microorganismos probióticos viables presentes. En este trabajo se analizaron seis productos adicionados con probióticos, con el fin de evaluar su concentración a lo largo de la vida útil del producto, se identificaron las cepas aisladas para corroborar la información declarada en la etiqueta y se determinó su perfil de susceptibilidad a antibióticos. Como resultado del análisis se encontró que las cepas adicionadas a los productos evaluados se mantienen en concentraciones aceptables durante los 28 días de vida útil de los productos. La identificación de las cepas aisladas no coincidió, en varios casos, con la estipulada por la etiqueta, no obstante, el método utilizado se basa en la capacidad de fermentar carbohidratos y no en pruebas genotípicas. Con respecto a los perfiles de susceptibilidad encontrados para las cepas analizadas, son necesarios estudios adicionales que establezcan la naturaleza intrínseca o adquirida de los determinantes de resistencia, y que evidencien si estos están codificados en elementos transferibles entre bacterias.
Palabras clave: Bacterias probióticas, perfil de sensibilidad a antibióticos, consumo humano.
In the last years, due to the high demand of food products supplemented with probiotics and the multiple nutritional and therapeutic benefits associated with them, research on these microorganisms has advanced considerably, including their selection and characterization. As a general recommendation, several entities as World Health Organization (WHO) and United Nations Organization for Agriculture and Food recommend that the specification of the alive species contained and their number shall appear in the label of the product. In the present study, six different commercially available products, supplemented with probiotics were analyzed, in order to evaluate the concentration of microorganisms through the shelf life of the product, identify the strains isolated and determine the antibiotic susceptibility pattern of these. Results demonstrated that the strains isolated kept acceptable concentrations during the 28 days of storage. Nevertheless, the identification of these strains variated from the one reported on the label on several of the products tested. This can be due to the commercial method used for the identifications, which is based in the carbohydrate fermentation pattern and not in genotypic trials. The antimicrobials’ susceptibility patterns found show that further research shall be performed in order to establish the intrinsic or acquired nature of the resistance determinants, and if these are codified by transferable elements among bacteria.
Key words: Probiotic bacteria, antimicrobial sensibility pattern, human consumption.
Facultad de Microbiología, Universidad de Costa Rica, Centro de Investigación en Enfermedades Tropicales. Costa Rica
El término probiótico se utiliza para definir a aquellos microorganismos que, al ser ingeridos vivos y en cierto número, proveen beneficios para la salud más allá de la nutrición básica inherente (1); siendo las bacterias ácido lácticas el grupo de organismos más comúnmente utilizado bajo este concepto (2).
El efecto de las bacterias probióticas en productos lácteos fermentados es bastante complejo, ya que no solo contribuyen con la preservación del alimento debido a la producción de ácido láctico y otros compuestos antimicrobianos, sino que también aportan compuestos responsables del aroma y sabor (acetaldehído entre otros) y polisacáridos extracelulares que le dan al producto características organolépticas deseables e incrementan su valor nutricional mediante la liberación de aminoácidos y vitaminas (2). Adicional a esto, varios autores describen beneficios potenciales de tipo terapéutico asociados a su consumo, principalmente atribuibles a la alteración favorable que sufre la flora gastrointestinal por parte de estas bacterias (2). Dentro de estos beneficios se encuentra la competencia con bacterias patógenas por el sitio de unión, la reducción de la intolerancia a la lactosa, la disminución de la diarrea producida por rotavirus y la inducida por antibióticos, la prevención del cáncer de colón y la estimulación del sistema inmune, entre otros. (2).
Con el incremento en el interés de la comunidad científica por el estudio y conocimiento de los mecanismos de acción de los microorganismos probióticos, también han surgido dudas con respecto a su introducción en la cadena alimentaria, ya que eventualmente estos microorganismos podrían funcionar como reservorios de genes de resistencia que podrían ser transferidos a la flora comensal de animales y humanos y a las bacterias patógenas que residan temporalmente en el hospedero (3-4), razón por la cual es de suma importancia el verificar que las bacterias ácido lácticas a utilizar como probióticos carezcan de elementos de resistencia antes de ser adicionadas a productos alimenticios (5).
De la misma manera, como parte del proceso de certificación de la calidad de los productos adicionados con probióticos, la FAO recomienda que tanto la especie, así como la cantidad de microorganismos probióticos viables al final de la vida útil del producto, sean declarados en la etiqueta del mismo (6,7). No obstante, varios análisis microbiológicos en productos lácteos han demostrado que, en algunas ocasiones, no existe correspondencia entre la declaración de la etiqueta y la recuperación e identificación de bacterias obtenidas (3, 8).
Dado lo anterior, en el presente estudio se evaluó, cuantificó e identificó las bacterias probióticas presentes en productos lácteos de consumo frecuente en Costa Rica y se estableció su perfil de sensibilidad a antibióticos de uso común.
Las cepas de cultivos probióticos evaluadas fueron obtenidas a partir de muestras de seis diferentes productos (yogur clásico, yogur líquido, yogur de leche de cabra, yogur de soya, yogur suizo y leche agria) distribuidos en el comercio costarricense y que, según su declaración, contienen probióticos adicionados.
Para evaluar la sobrevida de las bacterias en los productos se realizaron recuentos, por triplicado, cada cuatro días durante los 28 días de vida útil de los productos. Las cepas aisladas a partir de estos recuentos fueron identificadas y se determinó su perfil de susceptibilidad a diversos antibióticos.
Se pesaron 10g del producto y se homogenizaron con 90ml de agua peptonada estéril (APE) 0,1%. Seguidamente se realizaron diluciones decimales en APE 0,1 % y se depositaron en placas de agar MRS (Man, Rogosa y Sharpe) 0,1ml de las diluciones y se esparcieron usando asa de Drigalski. Las placas fueron colocadas en jarras con atmósfera capnofílica y se incubaron por 4 días a temperatura ambiente. Una vez finalizado el periodo de incubación se procedió a contar las colonias con morfología característica (blancas y pequeñas) y a éstas se les realizó tinción de Gram y prueba de catalasa.
La identificación se realizó mediante el sistema API CHL50 de la casa distribuidora bioMérieux®, según las indicaciones del fabricante.
Para determinar el perfil de susceptibilidad a antibióticos de las cepas aisladas se utilizó un método de difusión en agar descrito por Zhou et al. (9). Brevemente, 200 μl de una suspensión de bacterias equivalente al McFarland 0,5 fue mezclada con 100 mL de agar MRS fundido. Se depositaron 25 mL en cuatro placas de petri estériles y una vez solidificado se colocaron sobre su superficie discos de penicilina (P, 10μg), cefalotina (KF, 30μg), cefuroxime (CXM, 30μg), ampicilina (AMP, 10μg), vancomicina (VA, 30μg), tetraciclina (TE, 30μg), eritromicina (E, 15μg), rifampicina (RD, 5μg), cloranfenicol (C, 30μg), gentamicina (CN, 10μg) y ciprofloxacina (CIP, 5μg). Se incubaron durante 48 horas a 37°C en atmósfera capnofílica. Una vez finalizado este periodo se llevó a cabo la lectura e interpretación de los resultados. Se utilizó como cepas de control Escherichia coli ATCC 25922 y Staphylococcus aureus ATCC 25923. La prueba se interpretó de acuerdo a las recomendaciones del CLSI para bacterias Gram positivas del año 2007 (10).
Se logró aislar e identificar diferentes cepas bacterianas a partir de cada producto evaluado. En la Tabla 1, se presenta la identificación lograda así como los microorganismos declarados en la etiqueta de cada lácteo estudiado.
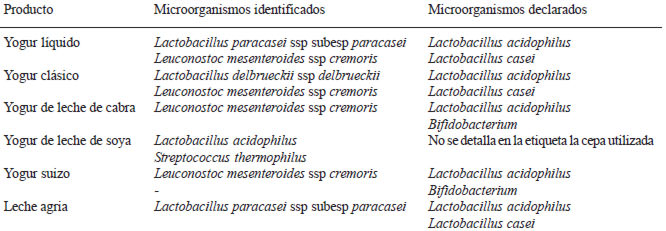
Con respecto a la identificación de los microorganismos aislados en este estudio, el sistema API CHL50 de bioMérieux® permite la identificación de bacterias lácticas mediante la determinación del perfil de fermentación de carbohidratos característico para cada bacteria.
En el caso de los bacilos Gram positivos aislados, las cepas de los diferentes productos fueron identificadas como L. delbrueckii para el yogur clásico y como L. paracasei para el yogur líquido y la leche agria. Es importante señalar que el fabricante establece que el sistema API CHL50 no es capaz de diferenciar entre las especies L. casei y L. paracasei por lo que es posible que las cepas identificadas como L. paracasei sí correspondan al cultivo probiótico declarado en la etiqueta.
Por otro lado, las cepas de cocos Gram positivos aisladas a partir de yogur líquido, clásico, de cabra y de soya fueron identificadas como Leuconostoc mesenteroides. Sin embargo, para estos productos no se reporta en la etiqueta la adición de ningún coco Gram positivo como cultivo probiótico.
A partir del yogur de leche de cabra, al igual que del yogur suizo únicamente se aíslan cepas de cocos y no se aíslan los microorganismos esperados (Bifidobacterium y L. acidophilus).
El yogur a base de soya no indica, en la etiqueta, cual es la bacteria utilizada como cultivo probiótico, pero a partir de este se aisló un bacilo Gram positivo el cual fue identificado como L. acidophilus. La cepa de cocos Gram positivos para este producto fue identificada mediante el API CHL50 como un Streptococcus thermophilus.
Por otro lado, los resultados de los recuentos de los seis productos durante los veintiocho días de su vida útil se mantienen entre 106 y 107 UFC/g para las cepas de bacilos. Los recuentos para los aislamientos de cocos Gram positivos se mantienen entre 107 y 108 UFC/g.
En cuanto a la susceptibilidad a los antibióticos de las cepas aisladas, se observó que en su mayoría presentan sensibilidad a los antibióticos evaluados en este estudio. Es importante destacar que todos los bacilos Gram positivos aislados mostraron resistencia a gentamicina y sólo uno de estos mostró sensibilidad a vancomicina. Por su parte, los cocos Gram positivos mostraron, al igual que los bacilos, resistencia a gentamicina, algún grado de resistencia a ciprofloxacina (3 cepas) y resistencia intermedia a vancomicina y eritromicina (1 cepa).
Los últimos años han sido testigos de la fuerte expansión del mercado de probióticos y, paralelamente, del aumento en el número de investigaciones dirigidas hacia aspectos fundamentales y las aplicaciones de los probióticos.
Dado el significativo aumento en el consumo de alimentos funcionales, como usualmente se les llama a los productos que contienen probióticos a nivel mundial, es importante que estos sean correctamente etiquetados y que las cepas de probióticos estén bien documentadas en cuanto a seguridad y funcionalidad (3).
La etiqueta de los productos analizados en este estudio establece que cada uno de ellos está adicionado con probióticos y en su mayoría, se establece qué microorganismos han sido adicionados (a excepción de uno).
Los productos yogur líquido, clásico y leche agria reportan la presencia de L. acidophilus, no obstante éste no se logra aislar. Las razones por las que no se aisla este microorganismo no son claras. No obstante, Temermann y colaboradores (3), reportan una situación similar, donde se dio un crecimiento pobre en agar MRS para L. acidophilus en comparación con otras especies de lactobacilos. Por el contrario, otros estudios no reportan dificultades para aislar este microorganismo bajo condiciones de anaerobiosis (11,12) a pesar que algunos autores describen modificaciones en los medios de cultivo como la acidificación adicional con ácido acético para alcanzar específicamente un pH de 5.4 (12), la adición de un suplemento de HCl-cisteína al 0.05% o la incubación en aerobiosis con una atmósfera enriquecida con 5% de CO2 (12).
Por otro lado, tanto en yogur líquido como en leche agria se logró el aislamiento de L. casei. Es importante señalar que el fabricante establece que el sistema API CHL50 no es capaz de diferenciar entre las especies L. casei y L paracasei por lo que las cepas aisladas pueden corresponder con las declaradas en la etiqueta. No obstante, cabe destacar que del yogur clásico no se aisló esta bacteria.
En el yogur suizo y en el de leche de cabra se declara la adición de Bifidobacterium y L. acidophilus, no obstante, no se logra el aislamiento de ninguna de estas bacterias. Con respecto a la no detección de Bifidobacterium, una de las posibles razones que lo explicaría seria la metodología empleada, ya que no se utilizó atmósfera anaerobia para la incubación de estas muestras. Dado que las bifidobacterias son bacterias anaerobias, los productores que adicionan este microorganismo utilizan la sobredosificación o la encapsulación bacteriana con el fin de alargar la viabilidad de estas cepas en los productos finales. En el caso de los yogures analizados, las etiquetas no indican la utilización de ninguna de estas estrategias ni tampoco se empaca al vacío, por lo que la viabilidad de esta cepa probiótica podría ser cuestionada.
Por otra parte, el mantenimiento de la cadena de frío de estos productos debe ser óptima, de manera que las cepas probióticas adicionadas no pierdan su viabilidad (7), factor que también podría haber influido en la no detección de algunos de los microorganismos declarados en las etiquetas de los productos.
Todo lo anterior demuestra que, al igual que lo establecen otros investigadores como Zong y colaboradores (13), la caracterización de bacterias ácido lácticas mediante morfología celular, colonial y por pruebas bioquímicas resulta poco exacta y con una alta probabilidad se pueden encontrar cepas que presentan características intermedias o que son incapaces de expresar algunas de sus características en determinados ambientes o condiciones de cultivo. Por lo tanto, las pruebas genotípicas van a ser más ventajosas en la tipificación de bacterias ácido lácticas permitiendo a la vez evaluar la diversidad entre cepas, tal y como lo propone la FAO (6).
Por otro lado, los seis productos evaluados mantuvieron un recuento promedio entre 106 y 108 UFC/g para las cepas de bacilos y cocos Gram positivos, durante los veintiocho días de su vida útil. Estos valores se consideran aceptables al estar cercanos al número de UFC recomendados para que un efecto probiótico pueda ser llevado a cabo por parte de las bacterias (7).
Con respecto a las pruebas de susceptibilidad a los antibióticos de las bacterias aisladas, se encontró que todas fueron resistentes a la gentamicina, concordando con otros estudios donde se reporta la resistencia por parte de estas bacterias hacia los aminoglicósidos (14,15,16). Hummel y colaboradores (11) establecen que esta resistencia es de carácter intrínseco y es atribuida a la ausencia de transporte de electrones mediado por citocromo.
Todas las cepas analizadas fueron sensibles a la eritromicina con excepción de una que resultó intermedia, coincidiendo con los trabajos de Zhou et al. (9), Katla et al (16) donde se describen bajos niveles de resistencia hacia este antimicrobiano.
Diversos autores reportan el aislamiento de lactobacilos con resistencia intrínseca hacia la vancomicina (3,15,17), lo cual concuerda con los resultados obtenidos en este estudio, donde la mayoría de las cepas de lactobacilos fueron resistentes a este antimicrobiano a excepción de la cepa identificada como L. acidophilus proveniente del yogur de soya. De igual manera, Temmerman y colaboradores (3) también reportan que un 35% de las cepas analizadas fueron sensibles a la vancomicina y Katla y colaboradores (15) reportan que ninguno de los aislamientos analizados por ellos de los géneros Lactobacillus, Lactococcus y Streptococcus presentó resistencia hacia vancomicina. Adicionalmente, Rojo y colaboradores (18) indican que algunas especies de Lactobacillus no poseen resistencia intrínseca a la vancomicina.
En cuanto a la resistencia a ciprofloxacina, se observa que una cepa, identificada como S. thermophilus proveniente del yogur de soya y las cepas de L. mesenteroides analizadas resultaron sensibles a dicho antibiótico. Al respecto, Katla y colaboradores (16) reportan una variabilidad natural en la susceptibilidad de estos géneros a la ciprofloxacina, mientras que Hummel y colaboradores (14) reportan que aproximadamente un 70% de las cepas analizadas por ellos fueron resistentes a la ciprofloxacina y clasifican esta resistencia como intrínseca.
No se encontró, en este estudio, resistencia a penicilina, tetraciclina ni a cloranfenicol, lo cual coincide con los estudios de Katla et al (16) y Zhou et al. (9), no obstante, algunos estudios reportan cepas de L. acidophilus y L. casei resistentes a penicilinas, gentamicina, tetraciclina, y cloranfenicol (18). Todas las cepas analizadas en este estudio fueron sensibles a rifampicina, sin embargo algunos investigadores han reportado resistencia hacia este antimicrobiano, la cual no se asocia a transferencia entre bacterias (15).
A manera de conclusión se puede establecer que los productos analizados presentan cantidades de bacterias lácticas en los niveles recomendados para ejercer su potencial efecto probiótico, no obstante, la identificación de estas cepas no es concluyente, son necesarios estudios adicionales, de carácter genético para alcanzar una identificación más exacta y poder corroborar la información del etiquetado. También, los perfiles de sensibilidad a antibióticos obtenidos no permiten categorizar las cepas evaluadas como seguras, se necesitan estudios más detallados capaces de determinar la naturaleza intrínseca o adquirida de los determinantes de resistencia a antimicrobianos así como que evidencien la capacidad de transferirse y en qué medida esto podría convertirse en un riesgo para la salud del consumidor.
Recibido: 12-01-2009
Aceptado: 21-03-2009